 2021.03.25
2021.03.25  5145次
5145次
01 MEGENA概要
基因共表达网络分析可以有效鉴定与复杂人类疾病相关的功能性共表达基因模块。最常用的共表达网络分析是加权基因共表达网络分析(WGCNA),它将复杂生物过程的基因共表达网络划分为高度相关的几个特征模块,其代表着几组高度协同变化的基因集,并可将模块与特定的临床特征建立关联,从中寻找发挥关键功能的基因,帮助识别参与特定生物学过程的潜在机制以及探索候选生物标志物。但是这种方法有一定局限性,无法在单个网络中共存不同级别的聚类,从而无法体现复杂网络的多尺度层次性。
为了解决这些缺点,MEGENA在拓扑领域采用了网络嵌入范式。换句话说,一个共同表达网络被嵌入在一个球面上,这样一个链接就不会与其他链接交叉。MEGENA通过:i)引入共表达相似性的质量控制,ii)并行化嵌入式网络构建,发展了一种新的聚类技术来识别平面滤波网络(PFNs)中的多尺度聚类结构。
MEGENA包括四个主要步骤:1)引入并行化、提前终止和先验质量控制的快速平面滤波网络构造(FPFNC);2)引入以分辨率参数为特征的模块结构紧凑性的多尺度聚类分析(MCA);3)采用多尺度中心分析(MHA)来识别高度复杂的网络聚类特征关联分析(CTA)探讨聚类与临床结果的相关性。图1显示了MEGENA的总体分析流程。
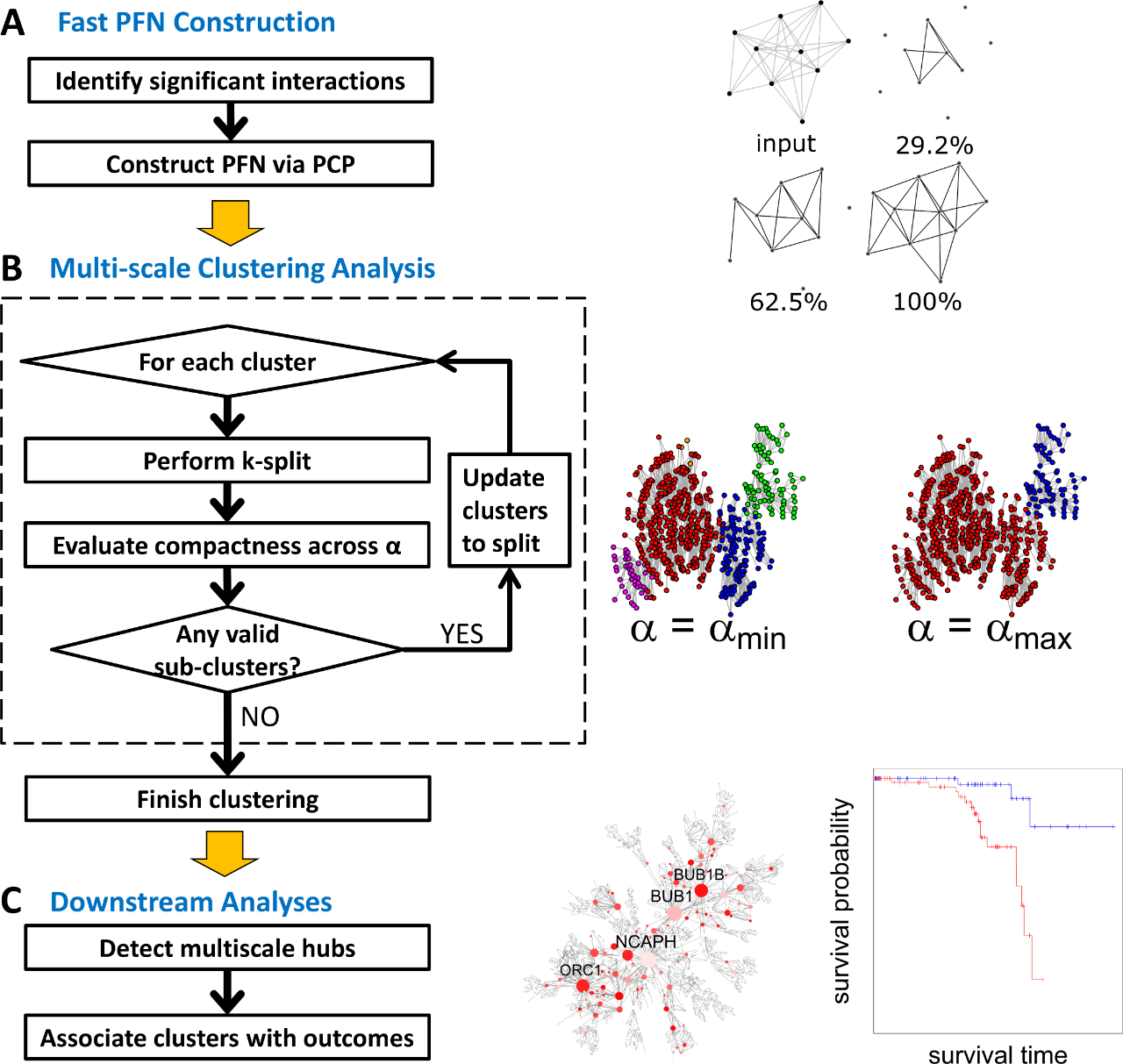
FPFNC主要遵循PMFG算法的网络嵌入原理来构造PFN。所有的基因对首先通过一个相似性度量进行排序,量化各自的相互作用强度,然后对其平面性进行迭代测试,以形成有利于包含具有较大相似性的基因对的嵌入式网络。为了使PFN构建在全基因组共表达网络分析中具有可扩展性,MEGENA开发了两种技术。首先,通过设置基因对相似性的错误发现率(FDR),在网络嵌入之前筛选掉不重要的交互作用。其次,提出了一种并行筛选程序(PCP)来提取更可能被嵌入的基因对子集,使得FPFNC能够更加高效地构建网络。
然后将FPFNC构造的PFN输入MCA,识别多尺度聚类。MCA结合了三个不同的准则来识别局部相干簇,同时保持全局最优分割。首先,利用最短路径距离(SPD)优化簇内紧密性。其次,利用局部路径索引(LPI)优化局部聚类结构。第三,采用整体模块化(Q)来确定最优划分。具体地说,MCA采用层次分裂法,通过k分裂和紧度估计两步迭代,将PFN中的复杂相互作用分解为不同分辨率的相干相互作用体。k-split通过优化SPD、LPI和Q来识别导致父网络最优划分的簇。
最后,利用MHA和CTA进行下游分析。MHA首先根据已建立的平面网络随机模型确定每个集群内的重要枢纽。在多个尺度上作为中心的节点称为多尺度中心。CTA通过主成分分析和相关分析评估个体聚类与临床结果的相关性。
02 MEGENA示例流程
1、计算相关性


2、计算PFN
在这一步中,平面滤波网络(PFN)是通过取显著相关对ijw来计算的。在使用不同的相似性度量的情况下,可以独立地将结果格式化为列名为c(“row”、“col”、“weight”)的3列数据帧,并确保weight列的范围在0到1之间。将此用作计算.PFN()也可以。



3、执行聚类
利用MCA聚类进行多尺度聚类分析。“MEGENA.output “是用于下游分析的核心输出,用于汇总和绘图。
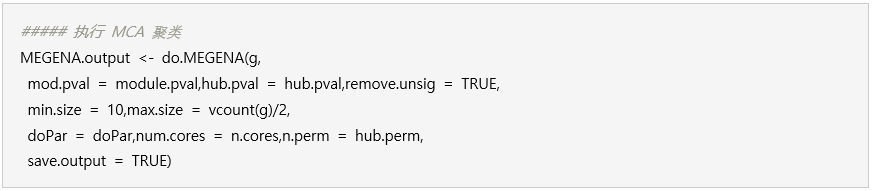
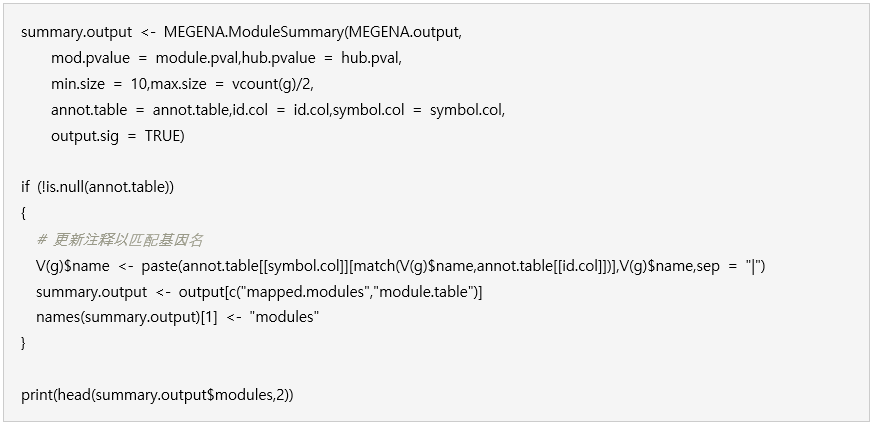
4、汇总结果



5、作图
可以选取感兴趣的网络模块作图




参考文献
[1] Zhang L, Peng TL, Wang L, Meng XH, Zhu W, Zeng Y, Zhu JQ, Zhou Y, Xiao HM, Deng HW. Network-based Transcriptome-wide Expression Study for Postmenopausal Osteoporosis. J Clin Endocrinol Metab. 2020 Aug 1;105(8):2678–91. doi: 10.1210/clinem/dgaa319. PMID: 32483604; PMCID: PMC7320836.
[2] Langfelder P, Horvath S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 2008 Dec 29;9:559. doi: 10.1186/1471-2105-9-559. PMID: 19114008; PMCID: PMC2631488.



 返回列表
返回列表